Anmerkung des Autors: Detaillierte Anleitung zur Erstellung von wissenschaftlichen Illustrationen wie Mechanismen, Signalwegen und Versuchsabläufen mit Nano Banana Pro – inklusive vollständiger Eingabeaufforderung-Vorlagen und API-Code. Über die APIYI-Plattform für nur 0,05 $ pro Bild.
Früher dauerte es Tage, eine Abbildung für ein SCI-Paper zu erstellen – entweder musste man einen Designer suchen oder mühsam selbst Grafiken in BioRender zusammenstellen. Heute kann Nano Banana Pro wissenschaftliche Mechanismus-Diagramme mit präzisen Beschriftungen direkt aus Textbeschreibungen generieren. Die Genauigkeit des Text-Renderings liegt bei über 97 % – Proteinnamen, Signalweg-Labels und zweisprachige Annotationen werden korrekt dargestellt, ohne Zeichensalat.
Kernwert: Nach der Lektüre dieses Artikels werden Sie die vollständigen Eingabeaufforderung-Vorlagen für fünf Kategorien wissenschaftlicher Illustrationen sowie den kompletten Code zur Batch-Generierung über die APIYI-API für nur 0,05 $ pro Bild beherrschen.

5 Szenarien für wissenschaftliche Illustrationen und Prompt-Vorlagen
Szenario 1: Zelluläre Signalweg-Mechanismen
Anwendungsbereiche: Zellbiologie, Molekularbiologie-Paper, Darstellung von Signalkaskaden und Protein-Phosphorylierungswegen.
Prompt-Vorlage:
Create a scientific mechanism diagram illustrating the MAPK/ERK signaling pathway.
Style: Clean white background academic illustration, suitable for SCI publication.
Layout: Vertical cascade flow from top (receptor) to bottom (nucleus).
Elements:
- Cell membrane at top with RTK receptor labeled "EGFR"
- Signal cascade: RAS → RAF → MEK → ERK with phosphorylation arrows
- Each protein shown as rounded rectangle with proper label
- Arrow styles: solid for activation, dashed for inhibition
- Nucleus at bottom with transcription factor labeled "ELK1"
Colors: Blue for membrane proteins, green for cytoplasmic kinases, red for nuclear targets
Text: All labels in English, 12pt equivalent, clearly legible
Resolution: 4K, aspect ratio 3:4
Vereinfachte deutsche Version des Prompts:
Erstelle ein Diagramm des MAPK/ERK-Signalwegs im Stil einer wissenschaftlichen Illustration für Fachzeitschriften, weißer Hintergrund.
Vertikale Anordnung von oben nach unten: Zellmembran (mit EGFR-Rezeptor) → RAS → RAF → MEK → ERK → Zellkern (ELK1).
Jedes Protein als abgerundetes Rechteck mit klarer Beschriftung, Aktivierungspfeile durchgezogen, Hemmungspfeile gestrichelt.
Blaue Membranproteine, grüne zytoplasmatische Kinasen, rote Kern-Transkriptionsfaktoren. 4K-Auflösung, 3:4 Format.
💡 Tipp: Bei molekularen Signalwegen empfiehlt es sich, die Proteinnamen jedes Knotens im Prompt explizit anzugeben. Nano Banana Pro rendert Fachbegriffe korrekt. Über APIYI (apiyi.com) kostet jedes 4K-Diagramm nur etwa 0,05 $, was die Erstellung mehrerer Varianten extrem kostengünstig macht.
Szenario 2: Experimentelle Flussdiagramme (Experimental Workflow)
Anwendungsbereiche: Methodik-Kapitel, Darstellung von Versuchsschritten, Probenverarbeitung oder Gerätebedienfolgen.
Prompt-Vorlage:
Design a scientific experimental workflow diagram for RNA-seq analysis pipeline.
Style: Professional flowchart, dark blue background (#0f172a), white and light blue elements.
Steps (left to right, 6 steps):
1. "Sample Collection" - test tube icon
2. "RNA Extraction" - spiral RNA icon
3. "Library Preparation" - fragment icons
4. "Sequencing" - chip/sequencer icon
5. "Bioinformatics Analysis" - computer/code icon
6. "Data Visualization" - chart icon
Each step: rounded rectangle box, icon above, step name below, connected by arrows
Color coding: wet lab steps in blue, computational steps in purple
Add percentage labels for typical time allocation at each step
4K resolution, 16:9 aspect ratio, publication quality
Szenario 3: Strukturillustrationen in der Zellbiologie
Anwendungsbereiche: Lehrmaterialien, Review-Artikel, Darstellung von Organellenstrukturen, Anatomie oder subzellulärer Lokalisation.
Prompt-Vorlage:
Create a detailed cell biology illustration showing mitochondrial structure and function.
Style: Scientific textbook illustration, clean white background.
Main elements:
- Cross-section view of mitochondrion (oval shape, 60% of frame)
- Outer membrane and inner membrane clearly labeled
- Cristae structure visible with labels
- ATP synthase complexes on inner membrane (labeled "ATP Synthase")
- Electron transport chain components (Complex I-IV) on inner membrane
- Matrix labeled with "TCA Cycle" and Krebs cycle intermediates
- Small arrows showing electron flow and proton gradient
- Legend box (bottom right): color key for different components
Text: All anatomical labels in English, bilingual caption below in Chinese and English
4K resolution, 4:3 aspect ratio
Wichtige Prompt-Technik: Verwenden Sie bei der Beschreibung von Zellstrukturen den Begriff Querschnittsansicht (cross-section view), um interne Strukturen besser sichtbar zu machen. Die Anforderung einer zweisprachigen Beschriftung (bilingual caption) ist besonders nützlich für internationale Publikationen.
Szenario 4: Infografiken zur Datenvisualisierung
Anwendungsbereiche: Ergebnispräsentation, Graphical Abstracts, Umwandlung komplexer Datenbeziehungen in intuitive visuelle Grafiken.
Prompt-Vorlage:
Create a scientific infographic summarizing CRISPR-Cas9 gene editing efficiency data.
Style: Modern scientific infographic, dark background (#1a1a2e), high contrast.
Layout: 2x2 grid of mini-charts:
Top-left: Bar chart "Editing Efficiency by Cell Type" - HEK293/iPSC/T-cell/Primary Neuron
Top-right: Pie chart "Off-target Distribution" - 3 categories with percentages
Bottom-left: Line graph "Efficiency Over Time (Days 1-14)" - two conditions
Bottom-right: Comparison table "sgRNA Design Methods" - 3 methods, 3 metrics
Color scheme: Blue (#3b82f6) for primary data, Green (#10b981) for positive results
All axis labels, titles, and data values visible and correctly spelled
Include figure title at top: "CRISPR-Cas9 Editing Performance Summary"
4K resolution, 16:9 aspect ratio
Szenario 5: Zweisprachige akademische Poster
Anwendungsbereiche: Akademische Konferenzen, Forschungspräsentationen, bei denen Inhalte professionell (z. B. Deutsch/Englisch oder Chinesisch/Englisch) präsentiert werden müssen.
Prompt-Vorlage:
Design a bilingual academic conference poster for a cancer immunotherapy study.
Style: Professional academic poster, dark navy background, white and gold accents.
Layout (top to bottom):
- Title bar: "Targeting PD-L1 in Triple-Negative Breast Cancer" (English)
"靶向 PD-L1 治疗三阴性乳腺癌" (Chinese, below English)
- Author line: "[Author Names] · [Institution] · [Conference Name] 2026"
- 3-column body:
Left: "Background / 研究背景" - 3 bullet points each language
Middle: Central mechanism diagram (simplified PD-1/PD-L1 pathway)
Right: "Results / 研究结果" - 2 key bar charts with bilingual labels
- Bottom: "Conclusion / 结论" banner with key finding
- QR code (bottom right) with caption "Scan for full paper"
Colors: Navy #0f172a background, gold #f59e0b accents, white text
All Chinese and English text must be correctly rendered and readable
4K resolution, 2:3 portrait aspect ratio (A0 poster format)
Schnelleinstieg in die Nano Banana Pro API für wissenschaftliches Zeichnen
Vorbereitung der Umgebung
pip install google-generativeai pillow
Minimalistisches Aufrufbeispiel (Erzeugung eines Mechanismus-Diagramms)
import google.generativeai as genai
import base64
# Aufruf über APIYI, Preis ca. 0,05 $/Bild (offiziell 0,24 $)
genai.configure(
api_key="YOUR_APIYI_KEY",
client_options={"api_endpoint": "vip.apiyi.com"}
)
model = genai.GenerativeModel("gemini-3-pro-image-preview")
prompt = """
Create a scientific MAPK/ERK signaling pathway diagram.
White background, academic publication style.
Show: EGFR → RAS → RAF → MEK → ERK → ELK1 cascade.
Each protein labeled clearly, arrows show activation/inhibition.
4K resolution, 3:4 aspect ratio, SCI journal quality.
"""
response = model.generate_content(
prompt,
generation_config=genai.GenerationConfig(
response_modalities=["IMAGE"],
resolution="4K",
aspect_ratio="3:4"
)
)
for part in response.candidates[0].content.parts:
if part.inline_data:
with open("mapk_pathway.png", "wb") as f:
f.write(base64.b64decode(part.inline_data.data))
print("Mechanismus-Diagramm generiert: mapk_pathway.png")
🚀 Schnellstart: Registrieren Sie sich bei APIYI (apiyi.com), um einen API-Key zu erhalten. Ersetzen Sie
YOUR_APIYI_KEYim obigen Code und führen Sie ihn aus. Die Plattform bietet kostenloses Testguthaben, sodass Sie Ihre erste Grafik innerhalb von 5 Minuten erstellen können.
Vollständigen Code für die Batch-Generierung anzeigen (inkl. mehrerer Szenarien, automatischer Benennung, Fehlerwiederholung)
import google.generativeai as genai
import base64
import os
import time
from typing import Optional
# Initialisierung des Clients - APIYI apiyi.com, 0,05 $/Bild
genai.configure(
api_key="YOUR_APIYI_KEY",
client_options={"api_endpoint": "vip.apiyi.com"}
)
model = genai.GenerativeModel("gemini-3-pro-image-preview")
def generate_scientific_figure(
prompt: str,
output_filename: str,
resolution: str = "4K",
aspect_ratio: str = "4:3",
max_retries: int = 3
) -> Optional[str]:
"""
Generiert wissenschaftliche Diagramme
Args:
prompt: Beschreibung der Grafik (Englisch funktioniert am besten)
output_filename: Name der Ausgabedatei (.png)
resolution: 1K / 2K / 4K
aspect_ratio: 16:9 / 4:3 / 3:4 / 1:1
max_retries: Anzahl der Wiederholungsversuche bei Fehlern
Returns:
Dateipfad bei Erfolg, None bei Fehler
"""
for attempt in range(max_retries):
try:
response = model.generate_content(
prompt,
generation_config=genai.GenerationConfig(
response_modalities=["IMAGE"],
resolution=resolution,
aspect_ratio=aspect_ratio
)
)
for part in response.candidates[0].content.parts:
if part.inline_data and part.inline_data.mime_type.startswith("image/"):
os.makedirs(os.path.dirname(output_filename) or ".", exist_ok=True)
with open(output_filename, "wb") as f:
f.write(base64.b64decode(part.inline_data.data))
return output_filename
except Exception as e:
print(f" Versuch {attempt + 1}/{max_retries} fehlgeschlagen: {e}")
if attempt < max_retries - 1:
time.sleep(5)
return None
# Liste vordefinierter Aufgaben für wissenschaftliche Grafiken
FIGURE_TASKS = [
{
"name": "MAPK-Signalweg",
"filename": "figures/fig1_mapk_pathway.png",
"resolution": "4K",
"aspect_ratio": "3:4",
"prompt": """
Scientific diagram: MAPK/ERK signaling pathway.
White background, academic style.
Top to bottom: EGFR receptor → RAS (GTP-bound) → RAF → MEK1/2 → ERK1/2 → nucleus (ELK1, c-Fos).
Each component: rounded rectangle, bold protein name label.
Activation arrows: solid black. Phosphorylation: circled 'P' symbol in red.
Color: blue membrane, green cytoplasm proteins, red nuclear targets.
Add "MAPK/ERK Signaling Pathway" title at top.
Publication quality, 4K.
"""
},
{
"name": "RNA-seq Workflow",
"filename": "figures/fig2_rnaseq_workflow.png",
"resolution": "4K",
"aspect_ratio": "16:9",
"prompt": """
Scientific workflow diagram: RNA-seq analysis pipeline.
Dark blue background, white icons and text.
6 steps left to right with connecting arrows:
1. Sample Collection (test tube icon)
2. RNA Extraction (helix icon)
3. Library Prep (fragment icon)
4. Sequencing (chip icon)
5. Bioinformatics (terminal icon)
6. Visualization (chart icon)
Steps 1-4 in blue (wet lab), steps 5-6 in purple (computational).
Each step: icon + step name + time estimate below.
Title: "RNA-seq Analysis Workflow". 4K, 16:9.
"""
},
{
"name": "Mitochondrien-Struktur",
"filename": "figures/fig3_mitochondria.png",
"resolution": "4K",
"aspect_ratio": "4:3",
"prompt": """
Scientific cell biology illustration: mitochondrion cross-section.
White background, textbook illustration style.
Detailed cross-section showing: outer membrane, inner membrane, cristae,
matrix with TCA cycle label, ATP synthase complexes (labeled) on inner membrane,
electron transport chain (Complex I-IV, labeled individually).
Small curved arrows showing proton gradient and electron flow.
Legend bottom-right: color key.
Bilingual labels: English primary, Chinese translation in parentheses.
Title: "Mitochondrial Structure and Function / 线粒体结构与功能".
4K, 4:3 ratio.
"""
},
]
def batch_generate_figures(tasks: list, output_dir: str = "figures") -> dict:
"""Batch-Generierung von wissenschaftlichen Grafiken"""
results = {"success": [], "failed": []}
total = len(tasks)
print(f"\nStarte Batch-Generierung von {total} Grafiken...")
print(f"Plattform: APIYI apiyi.com | Preis: 0,05 $/Bild | Erwartete Gesamtkosten: {total * 0.05:.2f} $\n")
for i, task in enumerate(tasks, 1):
print(f"[{i}/{total}] Generiere: {task['name']}")
path = generate_scientific_figure(
prompt=task["prompt"],
output_filename=task.get("filename", f"{output_dir}/figure_{i:02d}.png"),
resolution=task.get("resolution", "4K"),
aspect_ratio=task.get("aspect_ratio", "4:3")
)
if path:
results["success"].append({"name": task["name"], "path": path})
print(f" ✓ Fertig: {path}")
else:
results["failed"].append(task["name"])
print(f" ✗ Fehler: {task['name']}")
# Vermeidung von Rate-Limits
if i < total:
time.sleep(2)
print(f"\nGenerierung abgeschlossen: Erfolgreich {len(results['success'])}, Fehlgeschlagen {len(results['failed'])}")
print(f"Tatsächliche Kosten: {len(results['success']) * 0.05:.2f} $")
return results
if __name__ == "__main__":
results = batch_generate_figures(FIGURE_TASKS)
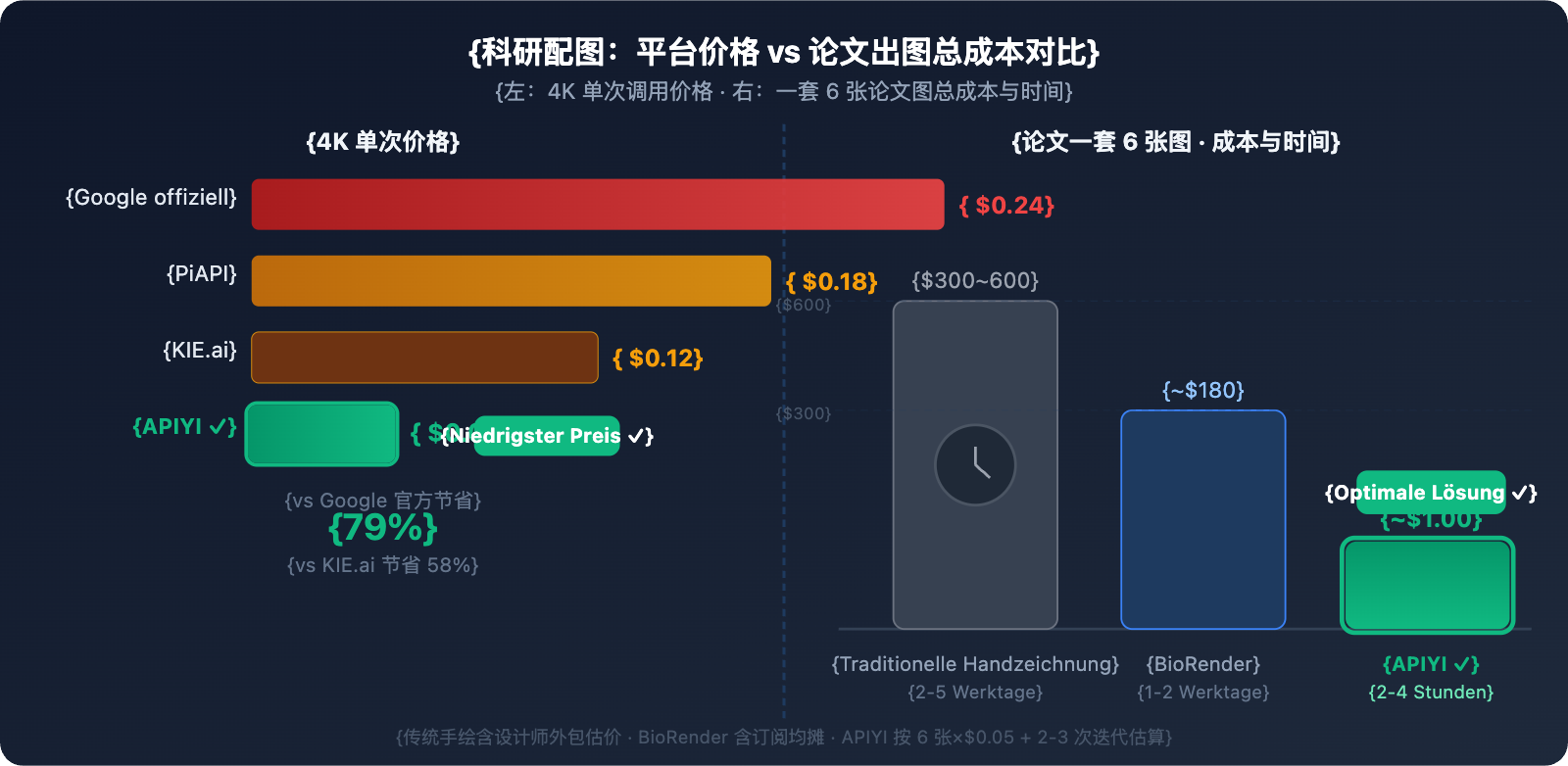
💰 Kostenvergleich: Die Erstellung von 20 wissenschaftlichen Grafiken (ein kompletter Satz Abbildungen für ein Paper) kostet bei Google offiziell etwa 4,80 $, über APIYI (apiyi.com) jedoch nur 1,00 $. Das entspricht einer Ersparnis von 79 %. Besonders in der Forschung, wo häufige Iterationen und Anpassungen nötig sind, ist dieser Kostenvorteil signifikant.
科研绘图提示词进阶技巧
提示词结构公式
优质科研图提示词 = 主题定义 + 风格规格 + 元素清单 + 标注要求 + 分辨率参数
| 要素 | 示例 | 作用 |
|---|---|---|
| 主题定义 | "Scientific diagram of CRISPR-Cas9 mechanism" | 告知模型专业领域 |
| 风格规格 | "White background, academic publication style" | 控制整体视觉风格 |
| 元素清单 | "Show: Cas9 protein, sgRNA, target DNA, PAM sequence" | 确保关键内容不遗漏 |
| 标注要求 | "All components labeled in English, bilingual Chinese caption" | 精准控制文字内容 |
| 分辨率参数 | "4K resolution, 16:9 aspect ratio" | 满足投稿规格 |
常见错误与修复
问题 1:文字标注位置错乱
❌ 错误提示: "label the proteins"
✅ 修复提示: "Each protein shown as rectangle with bold label text centered inside,
all labels: Arial 12pt equivalent, black text on white background"
问题 2:通路方向混乱
❌ 错误提示: "show the signaling cascade"
✅ 修复提示: "Top-to-bottom vertical flow: step 1 at top → step 2 → step 3 at bottom,
directional arrows pointing downward between each component"
问题 3:颜色缺乏区分度
❌ 错误提示: "use different colors for each component"
✅ 修复提示: "Color coding: membrane proteins #3b82f6 (blue), cytoplasmic #10b981 (green),
nuclear #f97316 (orange), inhibitory elements #ef4444 (red)"
不同学科场景的分辨率与宽高比推荐
科研图表的宽高比直接影响排版效果,以下是按投稿场景整理的推荐参数:
| 使用场景 | 推荐宽高比 | 推荐分辨率 | 说明 |
|---|---|---|---|
| 期刊单栏图 | 4:3 |
4K | Nature/Science 单列插图 |
| 期刊双栏图 | 16:9 |
4K | 跨栏宽幅复合图 |
| 竖版机制图 | 3:4 |
4K | 信号通路、树状图等纵向图 |
| 学术会议海报 | 2:3 |
4K | A0/A1 尺寸海报(竖版) |
| PPT/幻灯片配图 | 16:9 |
2K | 演讲演示用图 |
| 方形示意图 | 1:1 |
2K | 社媒发布或补充材料 |
| 横幅流程图 | 21:9 |
4K | 长流程实验步骤图 |
从提示词到发表:一套论文配图的完整工作流
以一篇细胞生物学论文为例,完整配图流程:
Step 1:规划图表结构(无 API 成本)
确定需要的图表类型和数量:
- Figure 1: 研究模型机制图(信号通路)→ 3:4 竖版
- Figure 2: 实验设计流程图 → 16:9 横版
- Figure 3A-C: 数据结果多面板图 → 16:9,2×3 布局
- Supplementary: 细胞结构示意图 × 2 → 4:3
共 6 张图,APIYI 总成本:$0.30
Step 2:逐张生成并迭代(每次 $0.05)
# 生成 → 检查 → 修改提示词 → 再生成
# 典型迭代次数:2-3 次/张,单图总成本 $0.10-0.15
Step 3:准确性核查(必须步骤)
对照 KEGG / UniProt / Reactome 数据库核查:
- 蛋白质名称拼写 ✓
- 通路方向和箭头含义 ✓
- 相互作用关系正确性 ✓
Step 4:格式处理
4K PNG → 期刊要求 300 DPI TIFF(使用 GIMP 或 Photoshop 转换)
如需矢量图:可将 PNG 导入 Adobe Illustrator 进行追踪
🎯 效率对比:传统手工绘制一套 6 张论文配图需 2-5 个工作日,使用 Nano Banana Pro + APIYI 整套流程约 2-4 小时,成本不到 $1。建议通过 APIYI apiyi.com 获取 API Key 后按本文模板直接套用,大幅压缩论文准备周期。
Plattformpreise und API-Vergleich
| Aufrufmethode | 4K-Einzelpreis | Stabilität | Formatunterstützung | Geeignete Szenarien |
|---|---|---|---|---|
| Google Offiziell | $0.24/Aufruf | ★★★★★ | Natives Format | Unternehmen mit hohem Budget |
| APIYI | $0.05/Aufruf | ★★★★★ | Natives Format ✅ | Batch-Generierung in der Forschung |
| KIE.ai | $0.12/Aufruf | ★★★☆☆ | Benutzerdefiniertes Format | Gelegentliche Nutzung |
| PiAPI | $0.18/Aufruf | ★★★★☆ | Benutzerdefiniertes Format | Kommerzielle Szenarien |
🎯 Empfehlung: Für Forscher und Labore erfordert die Überarbeitung von wissenschaftlichen Arbeiten oft wiederholte Anpassungen von Diagrammen, was einen hohen Bedarf an Batch-Generierung mit sich bringt. Es wird empfohlen, den Zugang über APIYI (apiyi.com) zu nutzen, um die Batch-Bilderstellung zu minimalen Kosten zu realisieren. Die Plattform unterstützt das native Google Gemini-Format, sodass keine Formatkonvertierung erforderlich ist und die direkte Kompatibilität mit allen Codebeispielen in diesem Artikel gewährleistet bleibt.
Häufig gestellte Fragen
F1: Können mit Nano Banana Pro erstellte wissenschaftliche Abbildungen direkt bei SCI-Fachzeitschriften eingereicht werden?
Ja, sie können verwendet werden, aber beachten Sie bitte die folgenden Punkte:
- Zwingende Genauigkeitsprüfung: KI-generierte Signalweg-Diagramme können Detailfehler aufweisen (z. B. ungewöhnliche Verzweigungen, fiktive Protein-Interaktionen). Vergleichen und validieren Sie diese vor der Einreichung unbedingt mit maßgeblichen Datenbanken (UniProt, KEGG, Reactome).
- Anforderungen an die Auflösung: Wählen Sie die 4K-Ausgabe. Die meisten Fachzeitschriften fordern eine Auflösung von mindestens 300 DPI, was durch 4K-Bilder voll erfüllt wird.
- KI-Deklaration: Top-Journals wie Nature und Science verlangen im Methodenteil eine Erklärung über die Verwendung von KI-Tools zur Erstellung von Illustrationen.
- Bedarf an Vektorgrafiken: Einige Zeitschriften verlangen die Bereitstellung von SVG/EPS-Vektorformaten. Nano Banana Pro gibt Rastergrafiken aus; für solche Zeitschriften ist eine zusätzliche Nachbearbeitung erforderlich.
Bilder, die über APIYI (apiyi.com) generiert werden, entsprechen exakt der offiziellen Qualität und eignen sich hervorragend für Preprints, Einreichungen bei Standard-Fachzeitschriften sowie Lehrmaterialien.
F2: Wie erstelle ich zusammengesetzte Abbildungen mit mehreren Panels (z. B. Figure 1A-1F)?
Nano Banana Pro unterstützt die Erstellung von Multi-Panel-Layouts in einem einzigen Bild. Die Eingabeaufforderung muss dies explizit angeben:
Create a multi-panel scientific figure (2 rows × 3 columns, labeled A-F):
Panel A (top-left): [Beschreibung des Inhalts von Bild A]
Panel B (top-center): [Beschreibung des Inhalts von Bild B]
Panel C (top-right): [Beschreibung des Inhalts von Bild C]
Panel D (bottom-left): [Beschreibung des Inhalts von Bild D]
Panel E (bottom-center): [Beschreibung des Inhalts von Bild E]
Panel F (bottom-right): [Beschreibung des Inhalts von Bild F]
Each panel: white background, bold letter label top-left (A, B, C...),
thin gray border between panels, consistent color scheme.
4K resolution, 16:9 aspect ratio.
Falls die Inhalte der Teilbilder stark variieren, können diese auch einzeln generiert und anschließend mit Python PIL zusammengefügt werden. Die Batch-Generierung von mehreren Teilbildern über APIYI (apiyi.com) kostet lediglich 0,30 $ (6 Bilder × 0,05 $).
F3: Chinesische Beschriftungen werden oft fehlerhaft oder unvollständig angezeigt. Wie lässt sich das lösen?
Die Rendering-Fähigkeiten von Nano Banana Pro für Chinesisch sind ausgezeichnet, erfordern jedoch die richtige Formulierung der Eingabeaufforderung:
- Sprache explizit deklarieren: Fügen Sie der Eingabeaufforderung hinzu:
"Chinese text must be correctly rendered in Simplified Chinese characters". - Schriftstil festlegen:
"Use standard Song/Hei font style for Chinese labels". - Textdichte kontrollieren: Verwenden Sie nicht zu viele chinesische Beschriftungen. Behalten Sie wichtige Markierungen bei und verschieben Sie sekundäre Informationen in die Legende.
- Zweisprachige Strategie: Englische Hauptbeschriftung + chinesische Ergänzung in Klammern, z. B.
"Mitochondria (线粒体)". Wenn Englisch dominiert, ist das Rendering des Chinesischen stabiler.
Falls das Problem weiterhin besteht, können Sie zuerst eine englische Version erstellen und dann über APIYI (apiyi.com) eine "Image-to-Image"-Anfrage stellen mit der Anweisung: "Ersetze auf Basis dieses Bildes alle englischen Beschriftungen durch Chinesisch".
Zusammenfassung
Die Kernpunkte für wissenschaftliches Zeichnen mit Nano Banana Pro:
- Textpräzision ist der größte Vorteil: Über 97 % Genauigkeit bei mehreren Sprachen. Proteinnamen und Signalweg-Labels werden präzise gerendert, was alle vergleichbaren Tools übertrifft.
- Spezielle Prompt-Strukturen für 5 Szenarien: Nutzen Sie das Template "Vertikale Kaskade" für Signalwege, "Horizontaler Prozessfluss" für Versuchsabläufe und definieren Sie bei zusammengesetzten Bildern klar das Panel-Layout.
- Preisvorteil durch APIYI: 0,05 $ pro Bild (statt 0,24 $ auf der offiziellen Website). Die Kosten für die Erstellung von Abbildungen für wissenschaftliche Arbeiten sinken um 79 %. Zudem wird der direkte Aufruf im Google-nativen Format unterstützt.
- Genauigkeit vor der Einreichung prüfen: KI-Bilder können fachliche Detailfehler enthalten. Eine Validierung anhand maßgeblicher Datenbanken ist unerlässlich.
Wir empfehlen, über APIYI (apiyi.com) einen automatisierten Workflow für wissenschaftliche Illustrationen aufzubauen. Speichern Sie die Codes aus diesem Artikel als Vorlagen und ersetzen Sie die Eingabeaufforderungen je nach Bedarf für die Batch-Generierung, um die Effizienz bei der Erstellung Ihrer Publikationen erheblich zu steigern.
Referenzen
-
Google Gemini API Bildgenerierungs-Dokumentation
- Link:
ai.google.dev/gemini-api/docs/image-generation - Beschreibung: Offizielle Parameterspezifikationen für Nano Banana Pro, einschließlich Best Practices für die Generierung wissenschaftlicher Bilder.
- Link:
-
Einführung in das Google PaperBanana Framework
- Link:
the-decoder.com/googles-paperbanana-uses-five-ai-agents-to-auto-generate-scientific-diagrams - Beschreibung: Technische Details zu Googles Multi-Agenten-Framework für die automatische Erstellung wissenschaftlicher Illustrationen.
- Link:
-
Bewertung von Nano Banana Pro für Anwendungen in den Life Sciences
- Link:
intuitionlabs.ai/articles/gemini-nano-banana-pro-life-sciences - Beschreibung: Systemleistungsbewertung für Szenarien in der Zell- und Molekularbiologie.
- Link:
-
Dokumentation für den APIYI-Plattformzugang
- Link:
docs.apiyi.com - Beschreibung: Informationen zum Erhalt von API-Keys, Anleitungen für den nativen Gemini-Format-Zugang und Preisdetails.
- Link:
Autor: Technik-Team
Technischer Austausch: Teilen Sie gerne Ihre wissenschaftlichen Illustrationen im Kommentarbereich. Weitere KI-Zeichentechniken finden Sie in der APIYI (apiyi.com) Technik-Community.