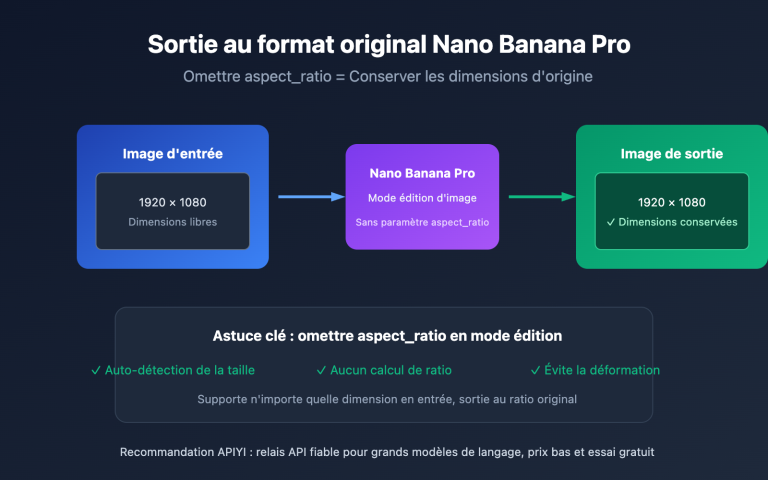
Note de l'auteur : Guide détaillé sur la manière d'utiliser Nano Banana Pro pour générer des schémas de mécanismes scientifiques, des cartes de voies métaboliques, des flux de travail expérimentaux et d'autres illustrations académiques. Inclut des modèles d'invites complets et du code d'appel API. Seulement 0,05 $ par image via la plateforme APIYI.
Dessiner une illustration pour un article SCI prenait autrefois des jours, entre la recherche d'un designer ou le bricolage manuel sur BioRender. Désormais, Nano Banana Pro peut générer directement des schémas de mécanismes scientifiques incluant des annotations précises à partir de descriptions textuelles — le taux de précision du rendu de texte atteint plus de 97 %. Les noms de protéines, les étiquettes de voies et les annotations bilingues sont tous rendus correctement, sans caractères illisibles.
Valeur ajoutée : À la fin de cet article, vous maîtriserez des modèles d'invites complets pour 5 types de scénarios de dessin scientifique, ainsi que le code complet pour générer en masse via l'API Nano Banana Pro sur APIYI à 0,05 $/image.

Capacités clés de Nano Banana Pro pour l'illustration scientifique
| Dimension de capacité | Performance | Valeur scientifique |
|---|---|---|
| Rendu de texte multilingue | Précision de 97%+, 12 langues | Noms de protéines et étiquettes de voies précis, sans caractères corrompus |
| Compréhension des annotations scientifiques | Entraînement approfondi sur la littérature académique et les manuels | Comprend les termes techniques comme MAPK, PI3K, etc. |
| Composition multi-panneaux | Supporte la mise en page complexe et la hiérarchie visuelle | Génère directement des visuels au format "Figure 1" pour vos articles |
| Sortie haute résolution | Trois niveaux : 1K / 2K / 4K | Répond aux exigences de qualité d'image pour la soumission aux revues |
| Incrustation de texte | Génération automatique de blocs d'annotation et de légendes | Plus besoin de passer par Photoshop pour rajouter du texte après coup |
Pourquoi choisir Nano Banana Pro pour l'illustration scientifique ?
Les graphiques scientifiques imposent des exigences extrêmement élevées aux outils de génération d'images par IA : il ne s'agit pas seulement de faire "beau", il faut surtout que les annotations soient précises, les termes techniques exacts et la hiérarchie des informations claire. C'est précisément là que réside l'avantage décisif de Nano Banana Pro par rapport aux autres outils.
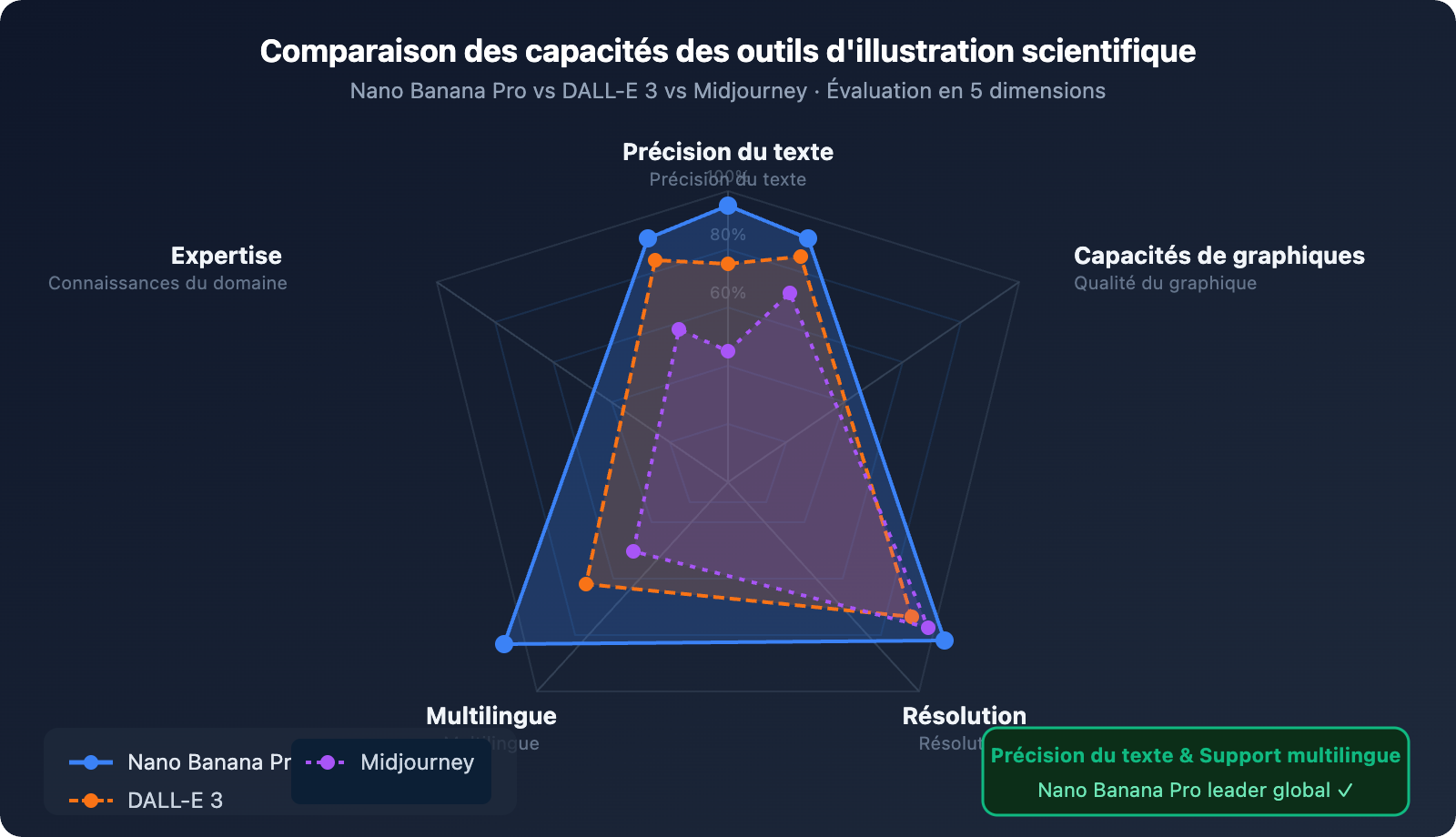
Comparaison avec des outils similaires : DALL-E 3 s'en sort un peu mieux sur le réalisme des schémas anatomiques, mais fait souvent des erreurs de texte lorsqu'il traite des figures composites avec beaucoup d'annotations ; Midjourney excelle dans le style artistique mais ses capacités d'annotation sont limitées ; Adobe Firefly est plutôt orienté vers le design commercial. Nano Banana Pro est actuellement le seul grand modèle de langage d'image à atteindre un tel niveau de performance simultanément dans le rendu de texte scientifique et la compréhension des connaissances spécialisées.
Google a également lancé le framework PaperBanana, basé sur ce même modèle. Il est conçu spécifiquement pour générer automatiquement des illustrations académiques à partir de descriptions méthodologiques. Testé systématiquement sur 584 échantillons lors de NeurIPS 2025, cela confirme son positionnement stratégique pour le monde de la recherche.
5 scénarios de dessin scientifique et modèles d'invites
Scénario 1 : Schéma des mécanismes des voies de signalisation cellulaire
Cas d'utilisation : Articles de biologie cellulaire ou moléculaire, présentation de cascades de signaux, voies de phosphorylation des protéines.
Modèle d'invite :
Create a scientific mechanism diagram illustrating the MAPK/ERK signaling pathway.
Style: Clean white background academic illustration, suitable for SCI publication.
Layout: Vertical cascade flow from top (receptor) to bottom (nucleus).
Elements:
- Cell membrane at top with RTK receptor labeled "EGFR"
- Signal cascade: RAS → RAF → MEK → ERK with phosphorylation arrows
- Each protein shown as rounded rectangle with proper label
- Arrow styles: solid for activation, dashed for inhibition
- Nucleus at bottom with transcription factor labeled "ELK1"
Colors: Blue for membrane proteins, green for cytoplasmic kinases, red for nuclear targets
Text: All labels in English, 12pt equivalent, clearly legible
Resolution: 4K, aspect ratio 3:4
Version simplifiée de l'invite (en chinois) :
绘制 MAPK/ERK 信号通路机制图,学术论文插图风格,白色背景。
从上到下纵向排列:细胞膜(含 EGFR 受体)→ RAS → RAF → MEK → ERK → 细胞核(ELK1)。
每个蛋白质用圆角矩形表示,标注清晰,激活箭头实线,抑制箭头虚线。
蓝色膜蛋白,绿色胞质激酶,红色核转录因子。4K 分辨率,3:4 比例。
💡 Conseil : Pour les schémas de voies moléculaires, il est recommandé de spécifier explicitement le nom de chaque protéine dans l'invite. Nano Banana Pro restituera correctement les termes techniques. En passant par APIYI (apiyi.com), chaque schéma 4K ne coûte que 0,05 $, ce qui rend la génération par lot de variantes extrêmement abordable.
Scénario 2 : Diagramme de flux expérimental (Workflow)
Cas d'utilisation : Chapitres méthodologiques, présentation des étapes expérimentales, processus de traitement des échantillons, séquences d'utilisation des instruments.
Modèle d'invite :
Design a scientific experimental workflow diagram for RNA-seq analysis pipeline.
Style: Professional flowchart, dark blue background (#0f172a), white and light blue elements.
Steps (left to right, 6 steps):
1. "Sample Collection" - test tube icon
2. "RNA Extraction" - spiral RNA icon
3. "Library Preparation" - fragment icons
4. "Sequencing" - chip/sequencer icon
5. "Bioinformatics Analysis" - computer/code icon
6. "Data Visualization" - chart icon
Each step: rounded rectangle box, icon above, step name below, connected by arrows
Color coding: wet lab steps in blue, computational steps in purple
Add percentage labels for typical time allocation at each step
4K resolution, 16:9 aspect ratio, publication quality
Scénario 3 : Illustration structurelle en biologie cellulaire
Cas d'utilisation : Matériel pédagogique, articles de revue (reviews), présentation de la structure des organites, anatomie tissulaire, localisation subcellulaire.
Modèle d'invite :
Create a detailed cell biology illustration showing mitochondrial structure and function.
Style: Scientific textbook illustration, clean white background.
Main elements:
- Cross-section view of mitochondrion (oval shape, 60% of frame)
- Outer membrane and inner membrane clearly labeled
- Cristae structure visible with labels
- ATP synthase complexes on inner membrane (labeled "ATP Synthase")
- Electron transport chain components (Complex I-IV) on inner membrane
- Matrix labeled with "TCA Cycle" and Krebs cycle intermediates
- Small arrows showing electron flow and proton gradient
- Legend box (bottom right): color key for different components
Text: All anatomical labels in English, bilingual caption below in Chinese and English
4K resolution, 4:3 aspect ratio
Astuce clé pour l'invite : Lors de la description de structures cellulaires, l'utilisation d'une vue en coupe (cross-section view) permet de mieux montrer l'organisation interne. L'ajout d'une légende bilingue (bilingual caption) permet une utilisation directe pour des soumissions dans des revues internationales ou locales.
Scénario 4 : Infographie de visualisation de données
Cas d'utilisation : Présentation de résultats, résumés graphiques, transformation de relations de données complexes en visuels intuitifs.
Modèle d'invite :
Create a scientific infographic summarizing CRISPR-Cas9 gene editing efficiency data.
Style: Modern scientific infographic, dark background (#1a1a2e), high contrast.
Layout: 2x2 grid of mini-charts:
Top-left: Bar chart "Editing Efficiency by Cell Type" - HEK293/iPSC/T-cell/Primary Neuron
Top-right: Pie chart "Off-target Distribution" - 3 categories with percentages
Bottom-left: Line graph "Efficiency Over Time (Days 1-14)" - two conditions
Bottom-right: Comparison table "sgRNA Design Methods" - 3 methods, 3 metrics
Color scheme: Blue (#3b82f6) for primary data, Green (#10b981) for positive results
All axis labels, titles, and data values visible and correctly spelled
Include figure title at top: "CRISPR-Cas9 Editing Performance Summary"
4K resolution, 16:9 aspect ratio
Scénario 5 : Poster académique bilingue
Cas d'utilisation : Conférences académiques, présentations de recherche nécessitant un affichage professionnel bilingue (anglais/chinois par exemple).
Modèle d'invite :
Design a bilingual academic conference poster for a cancer immunotherapy study.
Style: Professional academic poster, dark navy background, white and gold accents.
Layout (top to bottom):
- Title bar: "Targeting PD-L1 in Triple-Negative Breast Cancer" (English)
"靶向 PD-L1 治疗三阴性乳腺癌" (Chinese, below English)
- Author line: "[Author Names] · [Institution] · [Conference Name] 2026"
- 3-column body:
Left: "Background / 研究背景" - 3 bullet points each language
Middle: Central mechanism diagram (simplified PD-1/PD-L1 pathway)
Right: "Results / 研究结果" - 2 key bar charts with bilingual labels
- Bottom: "Conclusion / 结论" banner with key finding
- QR code (bottom right) with caption "Scan for full paper"
Colors: Navy #0f172a background, gold #f59e0b accents, white text
All Chinese and English text must be correctly rendered and readable
4K resolution, 2:3 portrait aspect ratio (A0 poster format)
Prise en main rapide de l'API de dessin scientifique Nano Banana Pro
Préparation de l'environnement
pip install google-generativeai pillow
Exemple d'appel minimaliste (générer un schéma de mécanisme)
import google.generativeai as genai
import base64
# 通过 APIYI 调用,价格 $0.05/张(官网 $0.24)
genai.configure(
api_key="YOUR_APIYI_KEY",
client_options={"api_endpoint": "vip.apiyi.com"}
)
model = genai.GenerativeModel("gemini-3-pro-image-preview")
prompt = """
Create a scientific MAPK/ERK signaling pathway diagram.
White background, academic publication style.
Show: EGFR → RAS → RAF → MEK → ERK → ELK1 cascade.
Each protein labeled clearly, arrows show activation/inhibition.
4K resolution, 3:4 aspect ratio, SCI journal quality.
"""
response = model.generate_content(
prompt,
generation_config=genai.GenerationConfig(
response_modalities=["IMAGE"],
resolution="4K",
aspect_ratio="3:4"
)
)
for part in response.candidates[0].content.parts:
if part.inline_data:
with open("mapk_pathway.png", "wb") as f:
f.write(base64.b64decode(part.inline_data.data))
print("机制图已生成:mapk_pathway.png")
🚀 Démarrage rapide : Inscrivez-vous sur APIYI (apiyi.com) pour obtenir votre clé API, copiez le code ci-dessus et remplacez
YOUR_APIYI_KEYpour l'exécuter. La plateforme offre des crédits de test gratuits, vous permettant de générer votre première figure scientifique en moins de 5 minutes.
Voir le code complet de génération par lot (incluant plusieurs scénarios, nommage automatique et gestion des erreurs)
import google.generativeai as genai
import base64
import os
import time
from typing import Optional
# 初始化客户端 - APIYI apiyi.com,$0.05/张
genai.configure(
api_key="YOUR_APIYI_KEY",
client_options={"api_endpoint": "vip.apiyi.com"}
)
model = genai.GenerativeModel("gemini-3-pro-image-preview")
def generate_scientific_figure(
prompt: str,
output_filename: str,
resolution: str = "4K",
aspect_ratio: str = "4:3",
max_retries: int = 3
) -> Optional[str]:
"""
生成科研图表
Args:
prompt: 图表描述(英文效果更佳)
output_filename: 输出文件名(.png)
resolution: 1K / 2K / 4K
aspect_ratio: 16:9 / 4:3 / 3:4 / 1:1
max_retries: 失败重试次数
Returns:
成功返回文件路径,失败返回 None
"""
for attempt in range(max_retries):
try:
response = model.generate_content(
prompt,
generation_config=genai.GenerationConfig(
response_modalities=["IMAGE"],
resolution=resolution,
aspect_ratio=aspect_ratio
)
)
for part in response.candidates[0].content.parts:
if part.inline_data and part.inline_data.mime_type.startswith("image/"):
os.makedirs(os.path.dirname(output_filename) or ".", exist_ok=True)
with open(output_filename, "wb") as f:
f.write(base64.b64decode(part.inline_data.data))
return output_filename
except Exception as e:
print(f" 尝试 {attempt + 1}/{max_retries} 失败: {e}")
if attempt < max_retries - 1:
time.sleep(5)
return None
# 预定义科研图表任务列表
FIGURE_TASKS = [
{
"name": "MAPK信号通路图",
"filename": "figures/fig1_mapk_pathway.png",
"resolution": "4K",
"aspect_ratio": "3:4",
"prompt": """
Scientific diagram: MAPK/ERK signaling pathway.
White background, academic style.
Top to bottom: EGFR receptor → RAS (GTP-bound) → RAF → MEK1/2 → ERK1/2 → nucleus (ELK1, c-Fos).
Each component: rounded rectangle, bold protein name label.
Activation arrows: solid black. Phosphorylation: circled 'P' symbol in red.
Color: blue membrane, green cytoplasm proteins, red nuclear targets.
Add "MAPK/ERK Signaling Pathway" title at top.
Publication quality, 4K.
"""
},
{
"name": "RNA-seq实验流程图",
"filename": "figures/fig2_rnaseq_workflow.png",
"resolution": "4K",
"aspect_ratio": "16:9",
"prompt": """
Scientific workflow diagram: RNA-seq analysis pipeline.
Dark blue background, white icons and text.
6 steps left to right with connecting arrows:
1. Sample Collection (test tube icon)
2. RNA Extraction (helix icon)
3. Library Prep (fragment icon)
4. Sequencing (chip icon)
5. Bioinformatics (terminal icon)
6. Visualization (chart icon)
Steps 1-4 in blue (wet lab), steps 5-6 in purple (computational).
Each step: icon + step name + time estimate below.
Title: "RNA-seq Analysis Workflow". 4K, 16:9.
"""
},
{
"name": "线粒体结构图",
"filename": "figures/fig3_mitochondria.png",
"resolution": "4K",
"aspect_ratio": "4:3",
"prompt": """
Scientific cell biology illustration: mitochondrion cross-section.
White background, textbook illustration style.
Detailed cross-section showing: outer membrane, inner membrane, cristae,
matrix with TCA cycle label, ATP synthase complexes (labeled) on inner membrane,
electron transport chain (Complex I-IV, labeled individually).
Small curved arrows showing proton gradient and electron flow.
Legend bottom-right: color key.
Bilingual labels: English primary, Chinese translation in parentheses.
Title: "Mitochondrial Structure and Function / 线粒体结构与功能".
4K, 4:3 ratio.
"""
},
]
def batch_generate_figures(tasks: list, output_dir: str = "figures") -> dict:
"""批量生成科研图表"""
results = {"success": [], "failed": []}
total = len(tasks)
print(f"\n开始批量生成 {total} 张科研图表...")
print(f"平台: APIYI apiyi.com | 单价: $0.05/张 | 预计总成本: ${total * 0.05:.2f}\n")
for i, task in enumerate(tasks, 1):
print(f"[{i}/{total}] 正在生成: {task['name']}")
path = generate_scientific_figure(
prompt=task["prompt"],
output_filename=task.get("filename", f"{output_dir}/figure_{i:02d}.png"),
resolution=task.get("resolution", "4K"),
aspect_ratio=task.get("aspect_ratio", "4:3")
)
if path:
results["success"].append({"name": task["name"], "path": path})
print(f" ✓ 完成: {path}")
else:
results["failed"].append(task["name"])
print(f" ✗ 失败: {task['name']}")
# 避免触发速率限制
if i < total:
time.sleep(2)
print(f"\n生成完毕: 成功 {len(results['success'])} 张,失败 {len(results['failed'])} 张")
print(f"实际成本: ${len(results['success']) * 0.05:.2f}")
return results
if __name__ == "__main__":
results = batch_generate_figures(FIGURE_TASKS)
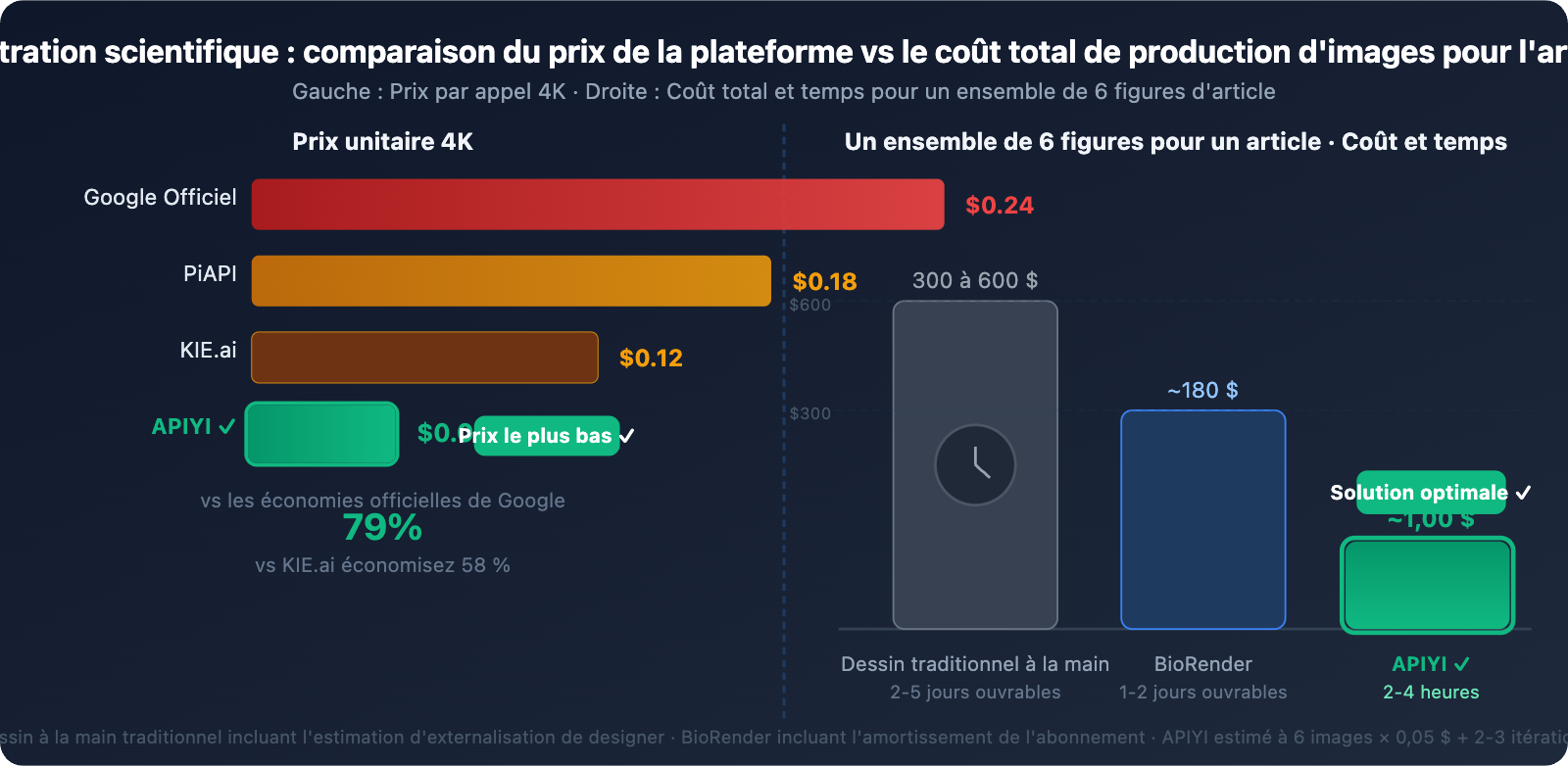
💰 Comparaison des coûts : Pour générer 20 illustrations scientifiques (un ensemble complet pour un article), le tarif officiel de Google est de 4,80 $, alors que via APIYI (apiyi.com), cela ne coûte que 1,00 $, soit une économie de 79 %. Pour les besoins de recherche nécessitant des itérations fréquentes, l'avantage économique est considérable.
Techniques avancées de prompts pour l'illustration scientifique
Formule de structure d'une invite
Une invite de qualité pour un schéma scientifique = Définition du sujet + Spécifications de style + Liste des éléments + Exigences d'étiquetage + Paramètres de résolution
| Élément | Exemple | Rôle |
|---|---|---|
| Définition du sujet | "Scientific diagram of CRISPR-Cas9 mechanism" | Informe le modèle du domaine d'expertise |
| Spécifications de style | "White background, academic publication style" | Contrôle l'aspect visuel global |
| Liste des éléments | "Show: Cas9 protein, sgRNA, target DNA, PAM sequence" | Garantit que le contenu clé n'est pas oublié |
| Exigences d'étiquetage | "All components labeled in English, bilingual Chinese caption" | Contrôle précisément le contenu textuel |
| Paramètres de résolution | "4K resolution, 16:9 aspect ratio" | Répond aux spécifications de soumission |
Erreurs courantes et corrections
Problème 1 : Placement désordonné des étiquettes de texte
❌ Mauvaise invite : "label the proteins"
✅ Invite corrigée : "Each protein shown as rectangle with bold label text centered inside,
all labels: Arial 12pt equivalent, black text on white background"
Problème 2 : Confusion dans la direction des voies de signalisation
❌ Mauvaise invite : "show the signaling cascade"
✅ Invite corrigée : "Top-to-bottom vertical flow: step 1 at top → step 2 → step 3 at bottom,
directional arrows pointing downward between each component"
Problème 3 : Manque de distinction par la couleur
❌ Mauvaise invite : "use different colors for each component"
✅ Invite corrigée : "Color coding: membrane proteins #3b82f6 (blue), cytoplasmic #10b981 (green),
nuclear #f97316 (orange), inhibitory elements #ef4444 (red)"
Recommandations de résolution et de format d'image par discipline
Le format d'image (aspect ratio) de vos schémas scientifiques influence directement la mise en page. Voici les paramètres recommandés selon le contexte de publication :
| Scénario d'utilisation | Format recommandé | Résolution recommandée | Description |
|---|---|---|---|
| Figure sur une colonne | 4:3 |
4K | Illustration standard pour Nature/Science |
| Figure sur deux colonnes | 16:9 |
4K | Schéma composite large traversant la page |
| Schéma de mécanisme vertical | 3:4 |
4K | Voies de signalisation, dendrogrammes, etc. |
| Poster de conférence | 2:3 |
4K | Posters de taille A0/A1 (format portrait) |
| Illustration PPT / Diapositive | 16:9 |
2K | Pour les présentations orales |
| Schéma carré | 1:1 |
2K | Réseaux sociaux ou matériel supplémentaire |
| Diagramme de flux en bandeau | 21:9 |
4K | Étapes expérimentales de long processus |
Du prompt à la publication : un workflow complet pour les illustrations de thèse
Prenons l'exemple d'un article en biologie cellulaire, voici le flux de travail complet :
Étape 1 : Planification de la structure (Coût API nul)
Déterminer le type et le nombre de schémas nécessaires :
- Figure 1 : Schéma du mécanisme du modèle (voie de signalisation) → Format 3:4 vertical
- Figure 2 : Flux de conception expérimentale → Format 16:9 horizontal
- Figure 3A-C : Panneau multi-résultats de données → Format 16:9, disposition 2×3
- Supplémentaire : Schémas de structures cellulaires × 2 → Format 4:3
Total de 6 images, coût total via APIYI : 0,30 $
Étape 2 : Génération et itération image par image (0,05 $ par essai)
# Générer → Vérifier → Modifier l'invite → Régénérer
# Nombre d'itérations typique : 2-3 fois par image, coût total par image 0,10-0,15 $
Étape 3 : Vérification de l'exactitude (Étape cruciale)
Vérifier par rapport aux bases de données KEGG / UniProt / Reactome :
- Orthographe des noms de protéines ✓
- Direction des voies et signification des flèches ✓
- Exactitude des relations d'interaction ✓
Étape 4 : Traitement du format
PNG 4K → TIFF 300 DPI requis par la revue (conversion via GIMP ou Photoshop)
Pour du vectoriel : importer le PNG dans Adobe Illustrator pour une vectorisation dynamique
🎯 Comparaison d'efficacité : Là où le dessin manuel traditionnel d'une série de 6 illustrations pour un article prendrait 2 à 5 jours de travail, l'utilisation de Nano Banana Pro combinée à APIYI permet de boucler le processus en 2 à 4 heures pour un coût inférieur à 1 $. Nous vous conseillons d'obtenir une clé API sur apiyi.com et d'appliquer directement les modèles de cet article pour réduire considérablement votre cycle de préparation de manuscrit.
Comparaison des prix des plateformes et des API
| Mode d'appel | Prix unitaire 4K | Stabilité | Formats supportés | Cas d'utilisation |
|---|---|---|---|---|
| Google Officiel | 0,24 $/appel | ★★★★★ | Format natif | Gros budgets d'entreprise |
| APIYI | 0,05 $/appel | ★★★★★ | Format natif ✅ | Génération par lots (Recherche) |
| KIE.ai | 0,12 $/appel | ★★★☆☆ | Format personnalisé | Usage occasionnel |
| PiAPI | 0,18 $/appel | ★★★★☆ | Format personnalisé | Scénarios commerciaux |
🎯 Conseil de sélection : Pour les chercheurs et les laboratoires, la révision d'articles nécessite souvent d'ajuster les graphiques à plusieurs reprises, ce qui implique une forte demande de génération par lots. Il est recommandé de passer par APIYI (apiyi.com) pour réaliser ces tâches au coût le plus bas. La plateforme supporte le format natif Google Gemini, sans conversion nécessaire, et est directement compatible avec tous les exemples de code de cet article.
Questions fréquemment posées
Q1 : Les figures de recherche générées par Nano Banana Pro peuvent-elles être soumises directement à des revues SCI ?
C'est possible, mais il faut prêter attention aux points suivants :
- Vérification de l'exactitude indispensable : Les schémas de voies métaboliques générés par l'IA peuvent comporter des erreurs de détail (comme des branches anormales ou des interactions protéiques fictives). Avant la soumission, comparez impérativement le résultat avec des bases de données faisant autorité (UniProt, KEGG, Reactome).
- Conformité de la résolution : Choisissez la sortie 4K. La plupart des revues exigent plus de 300 DPI, ce que les images 4K satisfont largement.
- Déclaration d'utilisation de l'IA : Les revues prestigieuses comme Nature ou Science exigent de déclarer l'utilisation d'outils d'IA pour la génération d'illustrations dans la section "Méthodes".
- Besoin de graphiques vectoriels : Certaines revues demandent des formats vectoriels SVG/EPS. Nano Banana Pro produit des images raster (pixels) ; pour ces revues, un traitement supplémentaire sera nécessaire.
Les images générées via l'appel à l'API de APIYI (apiyi.com) sont d'une qualité strictement identique à la version officielle, ce qui les rend parfaitement adaptées aux prépublications, aux revues de rang intermédiaire et aux supports pédagogiques.
Q2 : Comment générer des figures composites multi-panneaux (ex: Figure 1A-1F) ?
Nano Banana Pro permet de créer des mises en page multi-panneaux dans une seule image. Votre invite doit être très explicite :
Create a multi-panel scientific figure (2 rows × 3 columns, labeled A-F):
Panel A (top-left): [Description du contenu de la figure A]
Panel B (top-center): [Description du contenu de la figure B]
Panel C (top-right): [Description du contenu de la figure C]
Panel D (bottom-left): [Description du contenu de la figure D]
Panel E (bottom-center): [Description du contenu de la figure E]
Panel F (bottom-right): [Description du contenu de la figure F]
Each panel: white background, bold letter label top-left (A, B, C...),
thin gray border between panels, consistent color scheme.
4K resolution, 16:9 aspect ratio.
Si le contenu de chaque sous-figure varie trop, vous pouvez aussi les générer séparément puis les assembler avec Python (bibliothèque PIL). Passer par APIYI (apiyi.com) pour générer un lot de sous-figures ne coûte que 0,30 $ (6 images × 0,05 $).
Q3 : Les annotations en chinois s’affichent mal ou sont incomplètes, comment résoudre ce problème ?
Bien que Nano Banana Pro soit performant pour le rendu du chinois, cela nécessite une rédaction d'invite rigoureuse :
- Déclarer explicitement la langue : Ajoutez dans votre invite :
"Chinese text must be correctly rendered in Simplified Chinese characters". - Spécifier le style de police :
"Use standard Song/Hei font style for Chinese labels". - Contrôler la densité du texte : Ne surchargez pas l'image d'annotations en chinois. Gardez les plus importantes et déplacez les informations secondaires dans la légende.
- Stratégie bilingue : Utilisez l'anglais comme annotation principale et le chinois entre parenthèses, par exemple
"Mitochondria (线粒体)". Le rendu du chinois est plus stable quand l'anglais domine la structure.
Si le problème persiste, générez d'abord la version anglaise, puis utilisez la fonction "Image-to-Image" via APIYI (apiyi.com) en demandant de "remplacer toutes les annotations anglaises de cette image par du chinois".
Résumé
Voici les points essentiels pour réussir vos illustrations scientifiques avec Nano Banana Pro :
- La précision du texte est l'atout majeur : Avec un taux de précision multilingue de plus de 97 %, les noms de protéines et les étiquettes de voies métaboliques sont rendus avec une exactitude supérieure à tous les outils concurrents.
- Des structures d'invites dédiées pour 5 types de scénarios : Utilisez le modèle "flux en cascade vertical" pour les voies de signalisation, le "flux d'étapes horizontal" pour les protocoles expérimentaux, et définissez clairement la mise en page pour les figures composites.
- Choisissez APIYI pour le prix : À 0,05 $ l'image (contre 0,24 $ sur le site officiel), le coût de vos illustrations pour un article chute de 79 %. Le service supporte les appels directs au format natif de Google.
- Vérifiez impérativement l'exactitude avant soumission : Les images générées par IA peuvent contenir des erreurs sur des détails techniques. Validez toujours le résultat par rapport à des bases de données de référence.
Nous vous recommandons de mettre en place un flux de travail automatisé via APIYI (apiyi.com). En sauvegardant les codes de cet article comme modèles, il vous suffira de remplacer les invites selon vos besoins pour générer vos figures en série et booster l'efficacité de vos publications.
Références
-
Documentation sur la génération d'images de l'API Google Gemini
- Lien :
ai.google.dev/gemini-api/docs/image-generation - Description : Spécifications officielles des paramètres de Nano Banana Pro, incluant les meilleures pratiques pour la génération d'images scientifiques.
- Lien :
-
Présentation du framework Google PaperBanana
- Lien :
the-decoder.com/googles-paperbanana-uses-five-ai-agents-to-auto-generate-scientific-diagrams - Description : Détails techniques du framework multi-agents de Google dédié à la génération automatique d'illustrations académiques.
- Lien :
-
Évaluation de Nano Banana Pro pour les applications en sciences de la vie
- Lien :
intuitionlabs.ai/articles/gemini-nano-banana-pro-life-sciences - Description : Évaluation des performances du système pour les scénarios de biologie cellulaire et moléculaire.
- Lien :
-
Documentation d'accès à la plateforme APIYI
- Lien :
docs.apiyi.com - Description : Obtention de clés API, instructions d'accès au format natif Gemini et détails sur les tarifs.
- Lien :
Auteur : Équipe technique
Échanges techniques : N'hésitez pas à partager vos créations graphiques scientifiques dans les commentaires. Pour plus d'astuces sur le dessin par IA, visitez la communauté technique d'APIYI sur apiyi.com.